KEGG数据库的使用

KEGG数据库的使用
B站up _Willow_Liu
KEGG数据库的基本构成
整合了基因组、化学和系统功能信息的综合性数据库
大致分为系统信息、基因组信息、化学信息和健康信息四大类
查询代谢途径、酶、基因、产物等,BLAST查询未知序列的代谢途径信息
常用子数据库
PATHWAY(代谢途径数据库) 查询各种代谢途径
BRITE(代谢通路及同源基因数据库) 查询酶和底物之间的关系,查询某种酶的同源基因
GENES(基因数据库) 查询不同的基因或基因组的信息
LIGAND(配体数据库) 查询反映猴子那个各种化合物的信息
KEGG Organisms列出各物种的代码、KEGG使用三个英文小写字母代表各个物种
PATHWAY基本使用
查询各种代谢途径
KEGG–>KEGG PATHWAY–>Carbohydrate

红色方框–反应中的酶
红色椭圆–反应中的化合物
箭头–反应的方向
虚线–此反应可通过中间产物与其他途径发生联系
选择菜单 中 Animals–Mammals–hsa Homo sapiens(human)
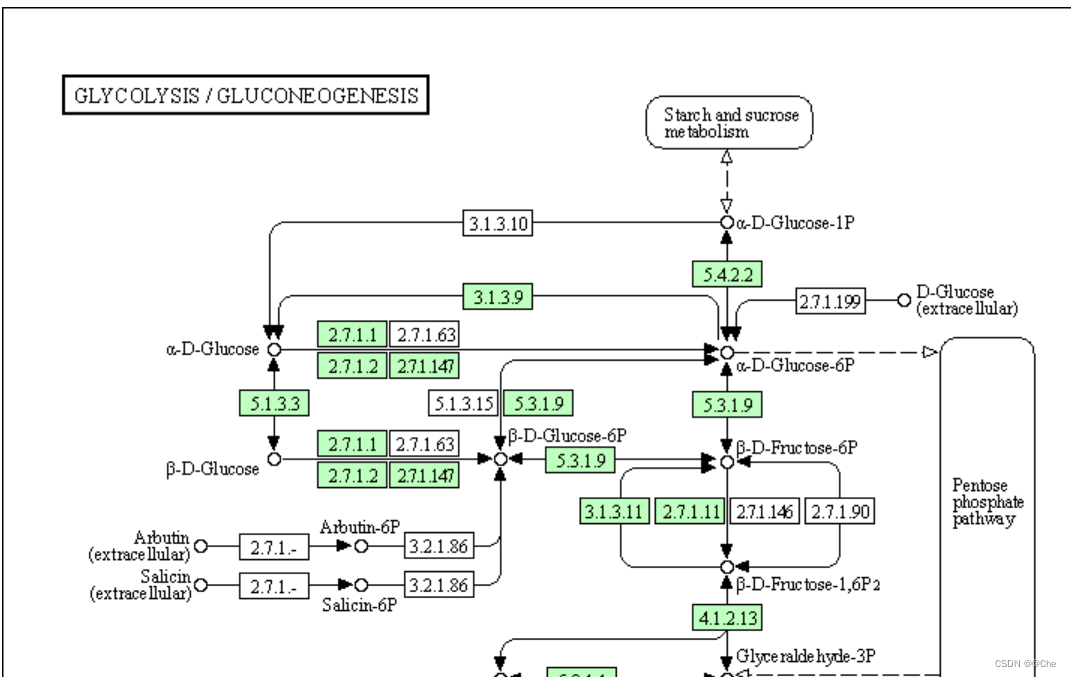
绿色框代表物种含有这种酶;点击该框弹出新的表单
BRITE数据库基本使用
用BRITE中的KO数据库查询某一基因的同源基因
例如查询柠檬酸合酶(gltA)的同源基因
1)进入BRITE数据库
2)点击Genes and proteins
3)在出现的页面上选择KEGG orthology(KO)
4)在左侧Search菜单栏输入gltA
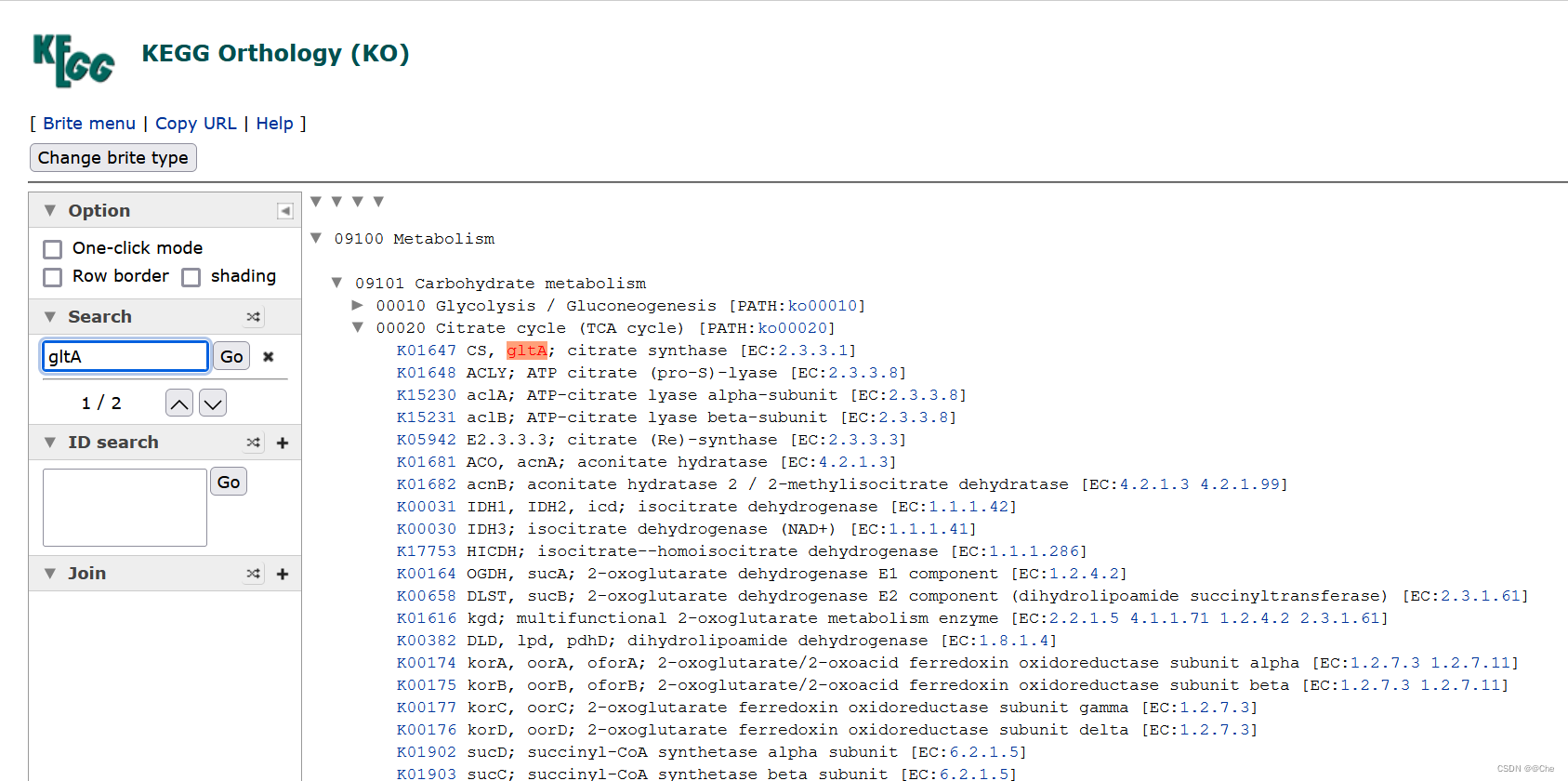
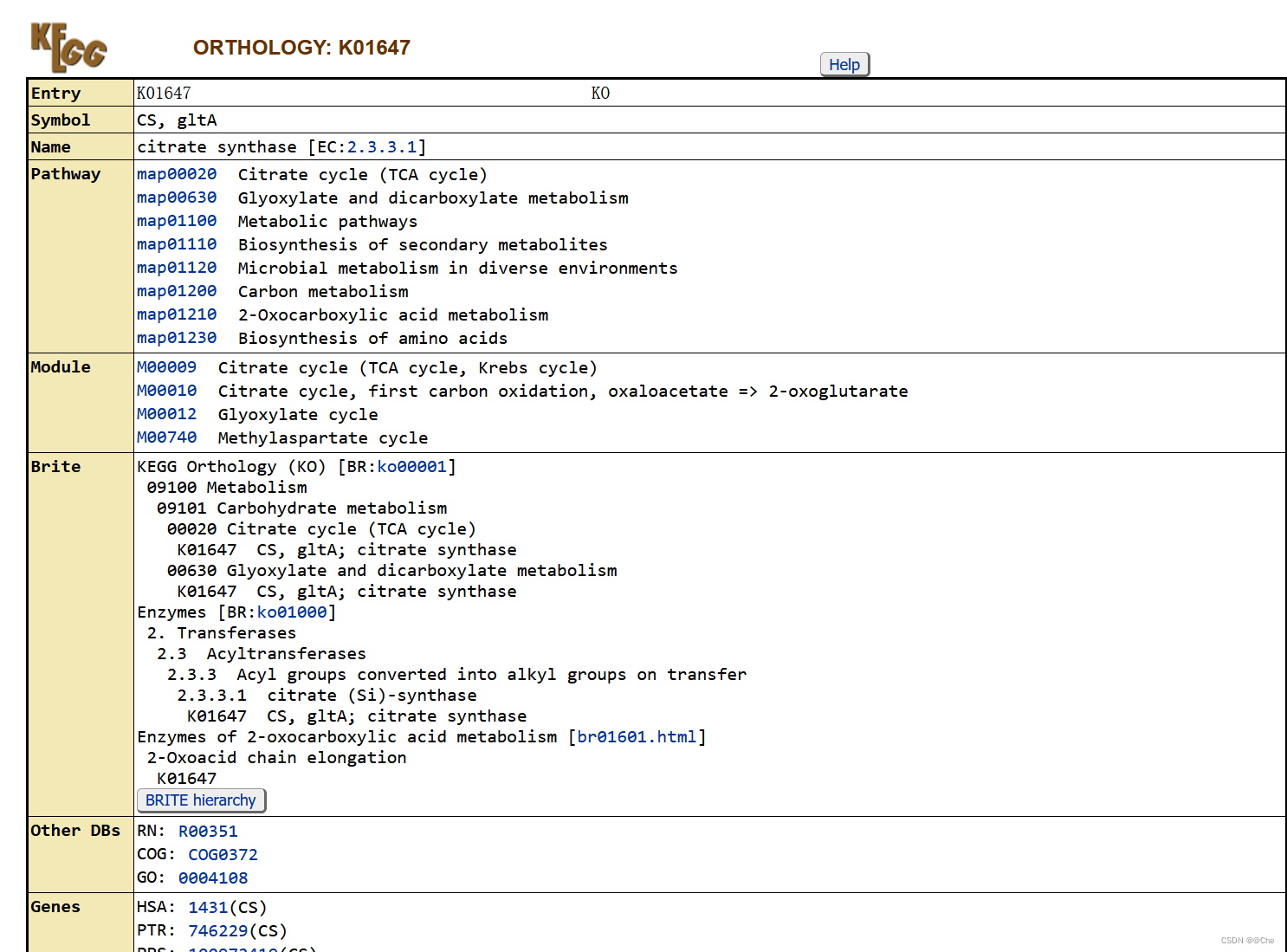
注意:基因的KO groups,KO是同源基因的分组,所以一个KO group包含不同代谢途径的多个基因。点击KO序号,即可查看此类同源基因
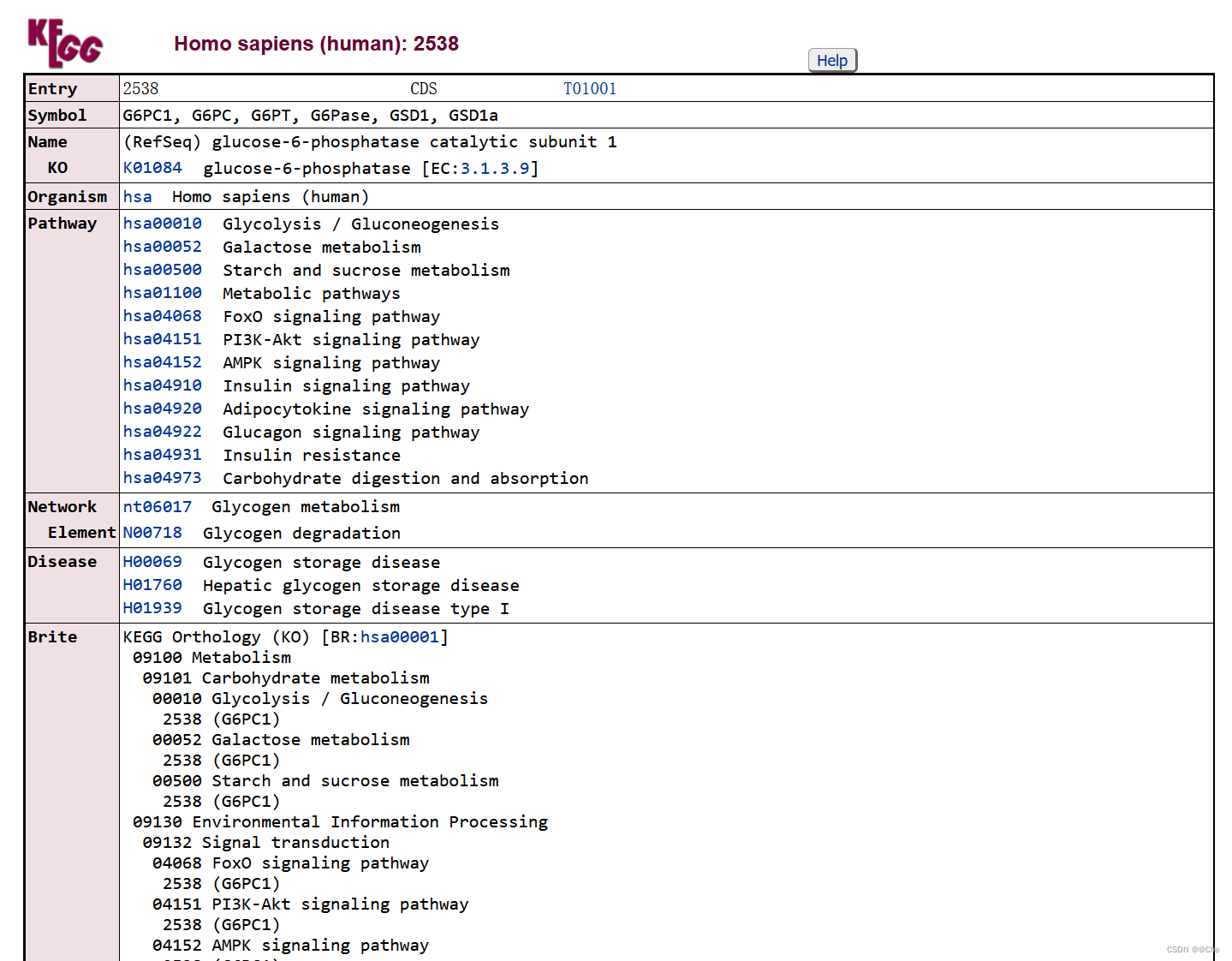
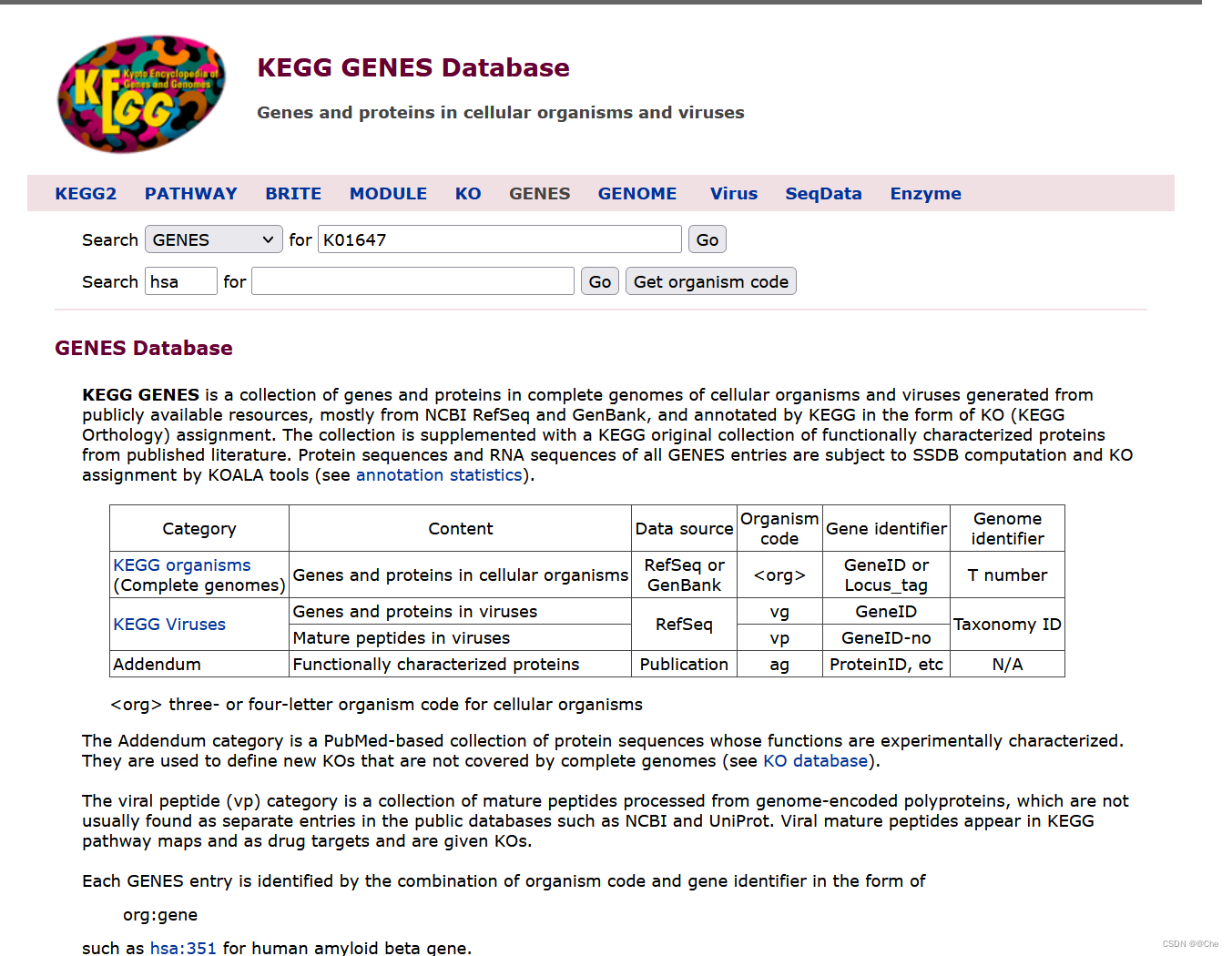
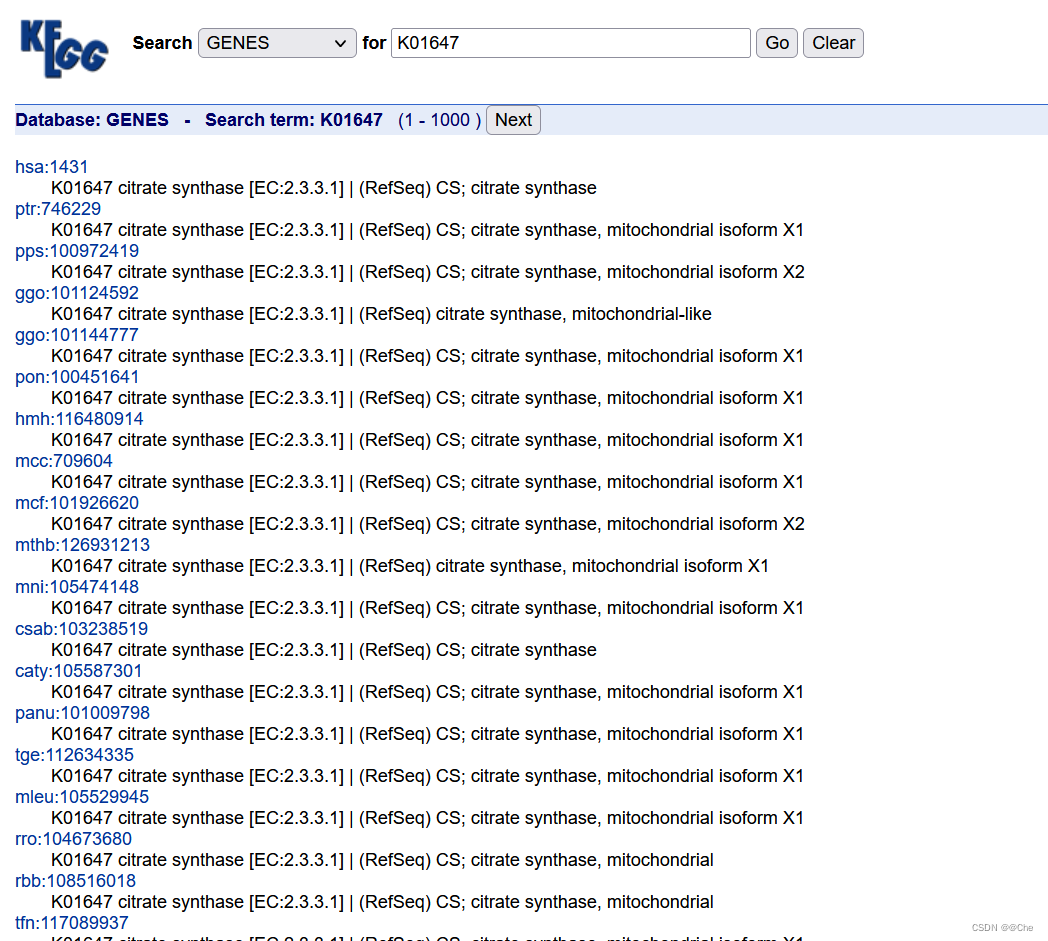
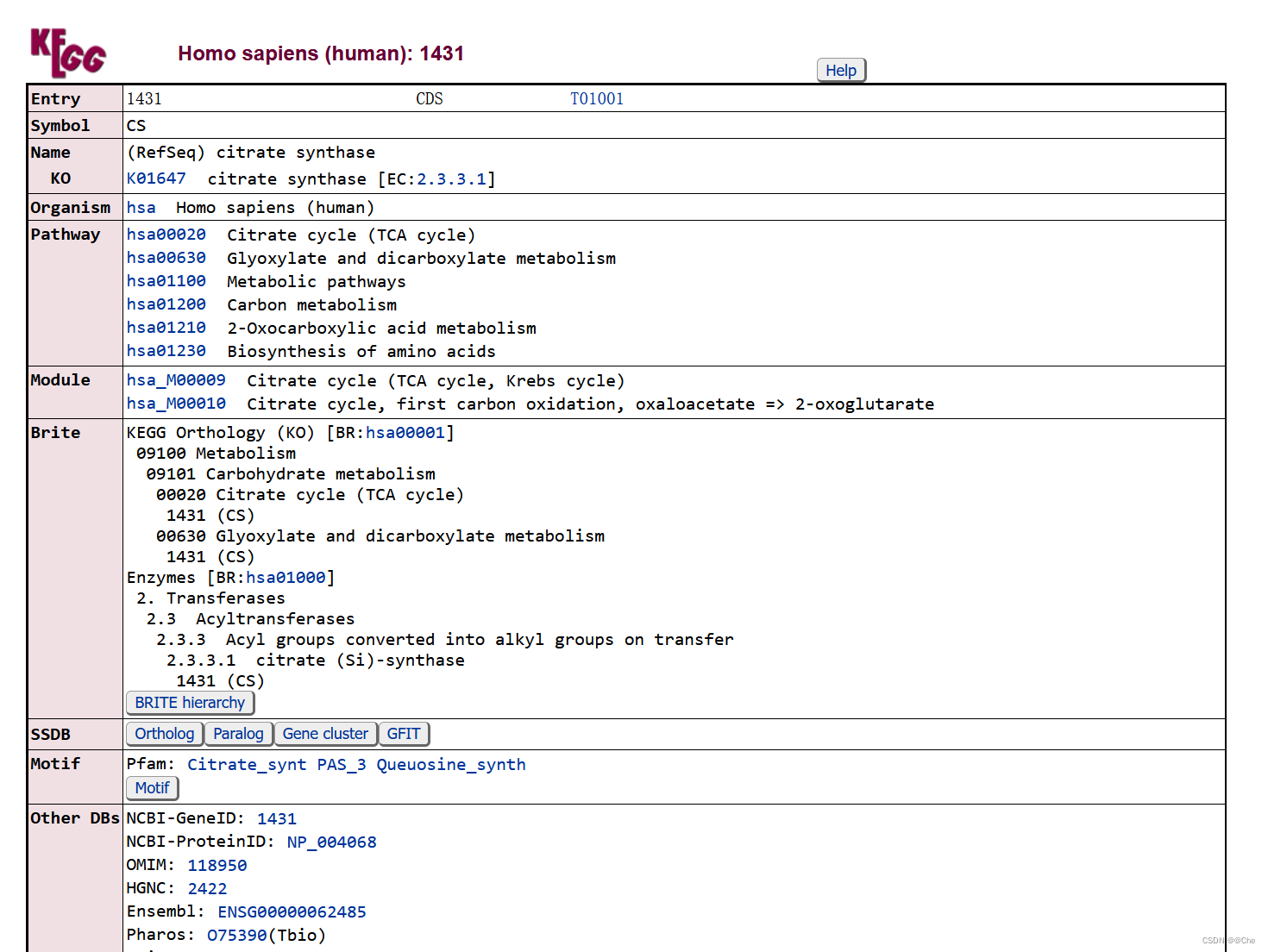
GENES数据库基本使用
查询不同的基因或基因组的信息
例如查询人类柠檬酸盐合成酶(gltA)基因具体信息
LIGAND数据库基本使用
查询化合物、化学反应或参与反应的酶
包含4个相关的子数据库

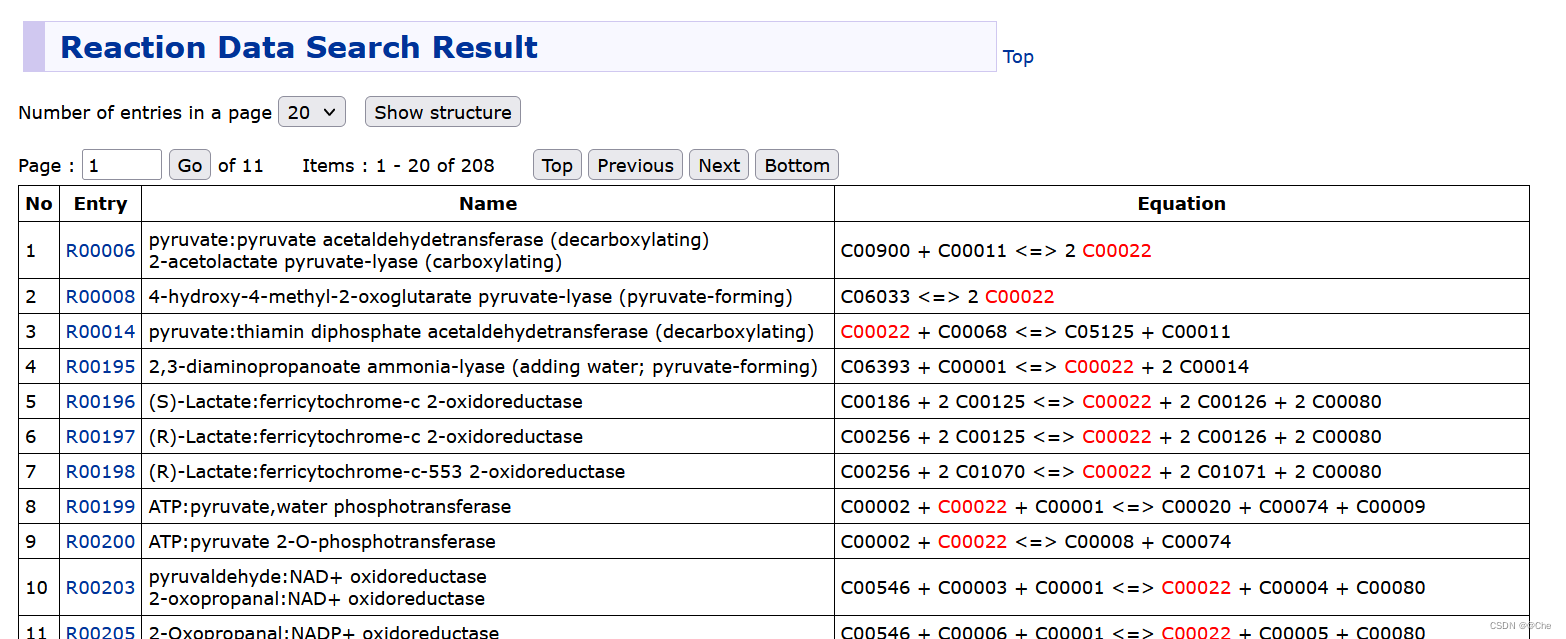
例如,查询数据库中Pyruvate(丙酮酸盐)信息
compound(复合物)中

reaction中
例如,人类的由Citrate到Acetyl-CoA的可能步骤
预测联系两种化合物的可能的反应途径
reaction中的反应路径搜索和预测中pathPred –>pathComp

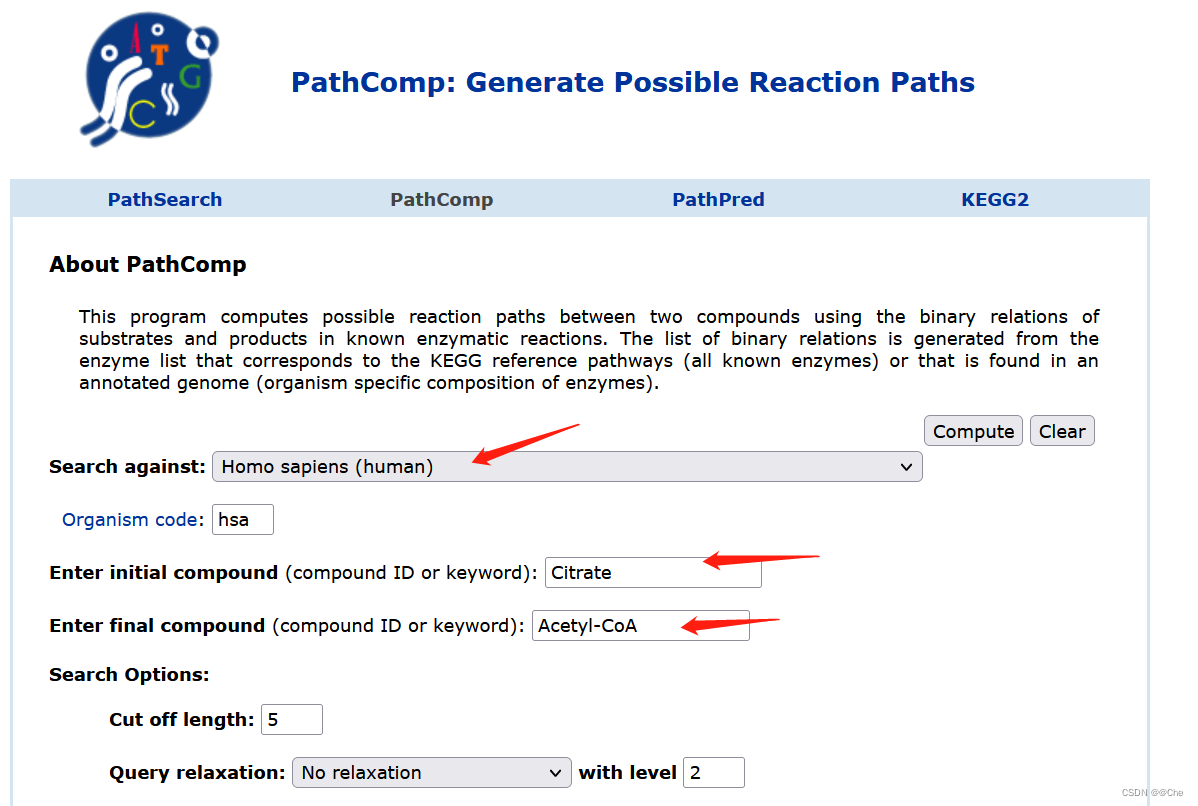
再按compute按钮
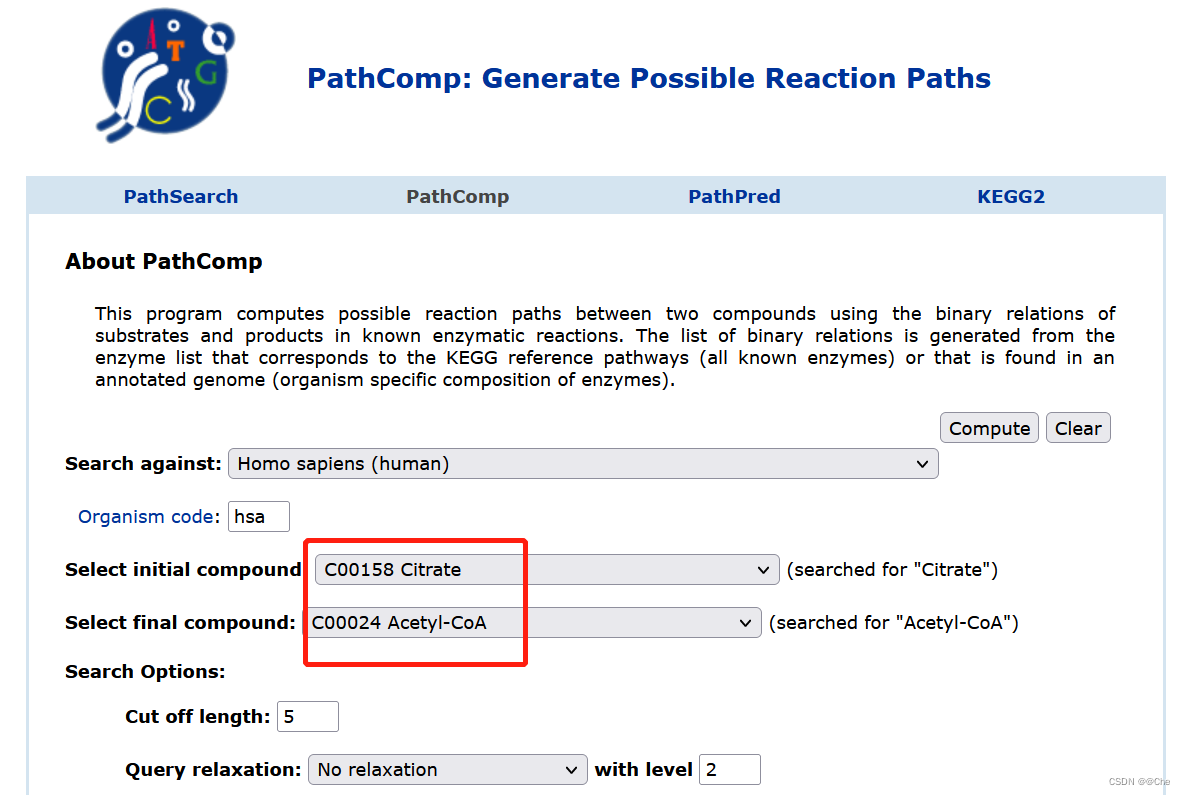
校准为库中的物质及编号 再按Compute按钮
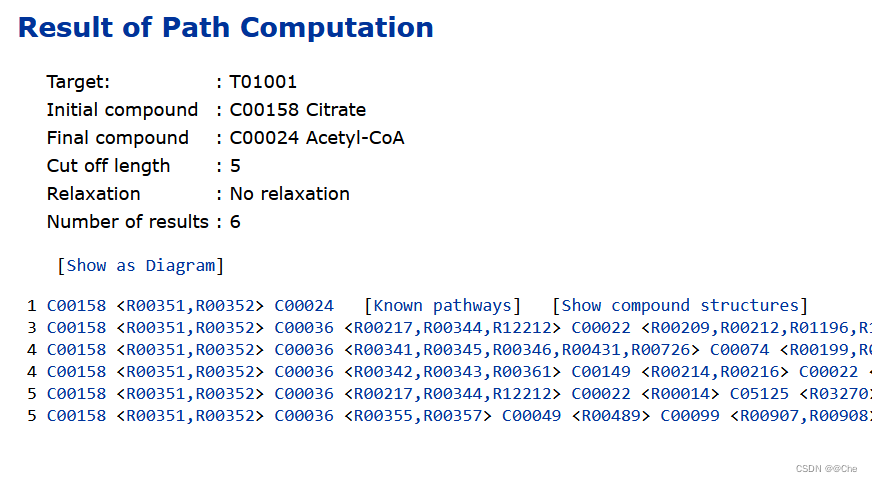
人类由Citrate到Acetyl-CoA的所有可能步骤
使用KEGG数据库对酶基因进行注释
作业
KEGG中查找MAPK信号通路(MAPK signaling pathway),简述其相关的生理功能,同时找出拟南芥中与植物激素相关的MAPK信号转导通路